研究理念
予期せぬ有害事象による市場撤退や, ドラッグリポジショニングの成功が示すように, 医薬品には開発者すら認識していない未知側面が多く存在する。我々は, そのような医薬品の「認識されていない側面」を理解と活用に向け, 生命科学データのパターン認識研究を進めている。医薬品の性質をパターンとしてレーダーチャートのように可視化することで, 医薬品の性質を既知・未知問わず俯瞰し, 認識可能となる。
俯瞰するための肝は, 解析対象を恣意性なく記述するデータの取得にある。真に恣意性のない数値化は困難だが, 恣意性を可能な限り軽減する方法論としてはどのようなものがあるだろうか。一つにオミクス解析など, 網羅性の活用が挙げられる。ある特定のレイヤーに属する変数を余さず取得することで, 少なくとも当該レイヤーに関しては解析対象の情報を恣意性なく数値化できる。特に医薬品の作用は解析対象医薬品への生体応答と解釈できるため, 解析対象医薬品を処理した生体を試料としたオミクスデータは, 医薬品の作用を記述する多変量として長らく用いられている。もう一つ恣意性を軽減する方法として, 画像データなど, 感覚器に近いデータの活用が挙げられる。取得に際し, 恣意性に大きく寄与するヒトの認識が介在しないデータを対象とすることで, 恣意性のない数値情報を得ることができる。一方, 画像や化合物構造のようなデータのうち, 生命科学的な意義を持つ情報は, 移動や回転に対し不変性を持つことが多く, そのまま扱うには冗長である。この点, 柔軟に帰納バイアスを導入可能な深層学習による潜在表現の抽出が有用である。潜在表現抽出の過程で恣意性が入ることにはなるが, 受け入れ性を考慮しながら設計可能な点は特筆に値する。
以上の考察のもと, 我々は医薬品に関する様々な生命科学データ (オミクスデータ, 化合物構造, 毒性病理画像, および科学論文) のパターン認識に取り組んでいる。
研究内容と教育
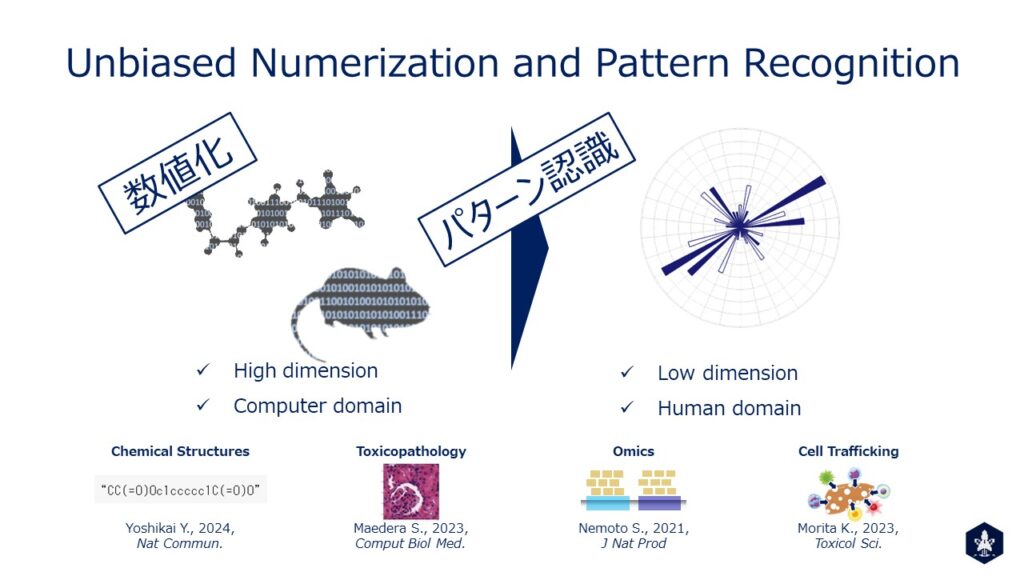
当グループではまず化合物を処理した培養細胞のトランスクリプトームデータの解析から, 上記戦略がin vitroでは成立することを確認した (Nemoto S., J Nat Prod., 2021)。そして現在では, in vivoやin humanなどより高位なレイヤーでの検討に取り組んでいます。具体的には, オミクスデータ (omics), 毒性病理画像 (image), 化合物構造 (structure), そして科学論文 (text)を対象に, 化合物の未知側面を引き出す方法論の開発と応用を進めている (Morita K., Toxicol Sci., 2023; Maedera S., Comput Biol Med., 2024; Yoshikai Y., Nat Commun., 2024)。恣意性のない数値化とパターン認識を軸とした一連のスキームは, ヒトの認識を拡張し, 知見として社会的な定着を促す点で汎用的である。哲学的な言葉を借用すれば, ヒトの処理能力を超えた感性(sense)で解析対象を扱い, ヒトが認識可能なパターンとしてから悟性(understanding)に基づき知見とするスキームと言える。そのため研究対象は化合物の未知側面理解に限らず, 多数の大学, 研究所, そして民間企業との共同研究も行っている。
バイオインフォマティクス, 及びケモインフォマティクス人材の育成にも力を入れている。薬学でデータサイエンスに取り組む最大の利点は, wetとsilico双方の視点が備わることにある。薬学では創薬という共通のゴールに向かい, 様々な専門家がチームを組んでタスクに取り組むため, そもそも学際的な土壌がある。学際的という言葉は時に専門性の不足といったネガティブな印象も与えますが, 双方に通じるからこそ見える世界観が基礎的にも応用的にも存在すると感じている。データを扱うのが得意で話のわかる便利屋さんではなく, 薬学と情報のダブルメジャーなればこその発見ができる研究者を目指したい人は, 気楽に連絡をください。
参考文献
- Lipton SA. Failures and successes of NMDA receptor antagonists: molecular basis for the use of open-channel blockers like memantine in the treatment of acute and chronic neurologic insults. NeuroRx. 2004 Jan;1(1):101-10. doi: 10.1602/neurorx.1.1.101.
- Nemoto S, Morita K, Mizuno T, Kusuhara H. Decomposition Profile Data Analysis for Deep Understanding of Multiple Effects of Natural Products. J Nat Prod. 2021 Apr 23;84(4):1283-1293. doi: 10.1021/acs.jnatprod.0c01381.
- Morita K, Mizuno T, Azuma I, Suzuki Y, Kusuhara H. Rat Deconvolution as Knowledge Miner for Immune Cell Trafficking from Toxicogenomics Databases. Toxicol Sci. 2023 Nov 6;197(2):121–31. doi: 10.1093/toxsci/kfad117.
- Maedera S, Mizuno T, Kusuhara H. Investigation of latent representation of toxicopathological images extracted by CNN model for understanding compound properties in vivo. Comput Biol Med. 2024 Jan;168:107748. doi: 10.1016/j.compbiomed.2023.107748.
- Yoshikai Y, Mizuno T, Nemoto S, Kusuhara H. Difficulty in chirality recognition for Transformer architectures learning chemical structures from string representations. Nat Commun. 2024 Feb 16;15(1):1197. doi: 10.1038/s41467-024-45102-8.